PhD Top Stories
Riccardo Sola
Caratterizzazione di peptidi antimicrobici ‘proline-rich’ nei cetacei: trovando nuovi potenziali antibiotici.
Corso di Dottorato in Biomedicina molecolare
Un uso incauto di antibiotici ha permesso la selezione e lo sviluppo di microbi ‘multi-farmaco resistenti’, che ormai causano migliaia di morti ogni anno nel mondo. Per superare le farmacoresistenze di tali microbi, servono urgentemente nuove classi di antibiotici. Nuovi antibiotici potrebbero arrivare dai peptidi antimicrobici o AMPs, promettenti molecole del sistema immunitario di molti animali e piante. La maggioranza di AMP uccide i batteri legando e distruggendone l’involucro protettivo, o membrana (AMP ‘membranolitici’). Gli AMP membranolitici sono efficaci contro molti batteri ma spesso tossici per le cellule umane, rompendo anche le loro membrane. Tuttavia, non tutti gli AMP funzionano in questo modo. I peptidi antimicrobici ricchi di prolina, o PrAMPs, non rompono la membrana batterica e sono solitamente meno tossici per le cellule umane rispetto ad altri AMP. I PrAMPs sfruttano alcune proteine trasportatrici quali SbmA come ‘cavalli di troia’ per entrare dentro i batteri; una volta dentro, legano e bloccano i ribosomi (le macchine molecolari che producono le proteine), uccidendo così i batteri. I ricercatori stanno tentando di ottimizzare i PrAMPs per estendere il loro raggio d’azione mantenendo la loro bassa tossicità. A questo scopo, lo studio di nuovi PrAMPs e la correlazione fra sequenze amminoacidiche e proprietà antibiotiche possono fornire indizi preziosi.
Il laboratorio del Prof. Scocchi studia da anni il PrAMP Bac7(1-35), derivato da un peptide prodotto da alcuni artiodattili (fra cui ovini e suini), e dotato di ottime proprietà antibiotiche. Usando come riferimento la sequenza amminoacidica di Bac7(1-35) abbiamo effettuato una ricerca bioinformatica di PrAMPs ‘equivalenti’ nei cetacei, che sono evolutivamente vicini agli artiodattili. La ricerca ha rivelato sette nuovi PrAMPs di cetacei (Fig.1) prodotti da sei specie diverse e con diverso grado di similarità a Bac7(1-35). Due peptidi chiamati Bal1 e Lip1, i più simili a Bac7(1-35) per sequenza amminoacidica, hanno mostrato attività antimicrobica più ampia e potente di Bac7(1-35) (Fig.1).
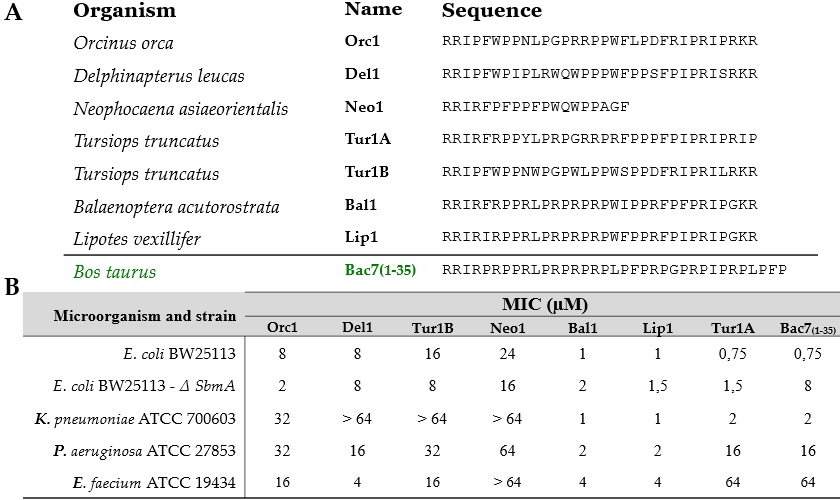
Figura 1: Sequenze (A) ed attività antimicrobica (B) dei PrAMP di cetaceo e Bac7(1-35).
la loro minima concentrazione inibente la crescita batterica (MIC) era = 4 µM contro molti batteri, inclusi Gram-negativi insidiosi come Pseudomonas aeruginosa e Gram-positivi come Enterococcus faecium. Inoltre, in assenza del trasportatore SbmA, Bal1 e Lip1 non calavano di efficacia come altri PrAMPs. Bal1 e Lip1 conservavano comunque un’ azione non-membranolitica, bloccante la sintesi proteica (Fig.2), come Bac7(1-35). Un dato interessante da notare è che un breve tratto della loro sequenza amminoacidica è conservato quasi identico in altri PrAMPs inibitori della produzione (o ‘sintesi’)proteica, suggerendo un ‘motivo di sequenza’ comune, correlato a questa proprietà. Anche a dosi ben superiori la MIC, Bal1 e Lip1 non causavano tossicità a globuli rossi e cheratinociti umani (non mostrato), causando solo tossicità verso le cellule leucemiche umane Mec-1 (Fig.2).
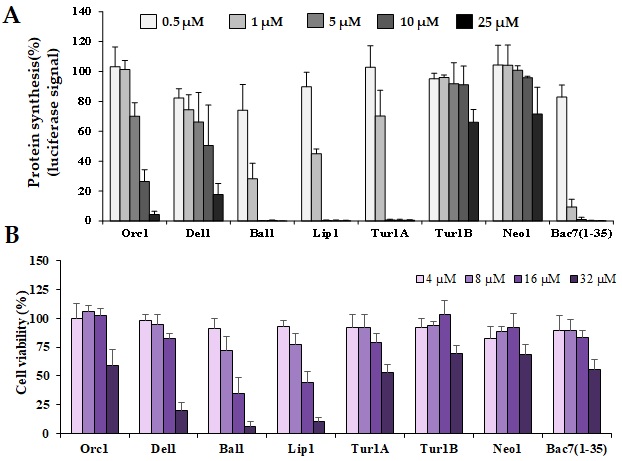
Figura 2: Effetti dei peptidi sulla sintesi proteica batterica (A) e sulla vitalità delle cellule umane MEC-1(B). A) misura della sintesi proteica tramite saggio di luciferasi, misurando la luminescenza di una proteina di riferimento (luciferasi). B) saggio di tossicità cellulare MTT. I risultati sono riportati come percentuale, rispetto al segnale di campioni non trattati coi peptidi.
Un secondo gruppo di PrAMPs (Orc1, Del1, Tur1B e Neo1) era abbastanza ‘atipico’, nel senso che non bloccava i ribosomi ma sembrava piuttosto danneggiare la membrana batterica (non mostrato). Nonostante il meccanismo d’azione, questi peptidi erano meno potenti e con raggio d’azione più ristretto di Bal1 e Lip1, ed alcuni non erano tossici nemmeno per la linea cellular MEC-1. Questi nuovi PrAMPs non comuni meritano sicuramente ulteriori studi. In conclusione, lo studio di questi nuovi PrAMPs di cetaceo ha rivelato sia peptidi promettenti che peptidi meno potenti ma ‘inusuali’. Sono stati identificati due PrAMPs con sequenze e meccanismo d’azione simili al già noto Bac7(1-35) ma più potenti e a più ampio raggio, ed altri peptidi membranolitici, non tossici per le cellule umane testate, ma meno efficaci di Bac7(1-35). Correlare le differenze nelle sequenze di questi PrAMPs alle differenze nelle loro proprietà può dare indizi utili alla progettazione razionale di PrAMPs ottimizzati, con maggiori probabilità di poter diventare nuovi antibiotici.
Autori ed affiliazioni
Riccardo Sola1, Mario Mardirossian2, Bertrand Beckert3 Laura Sanghez De Luna1 Dennis Prickett1 Alessandro Tossi1 Daniel N. Wilson3 Marco Scocchi12Dipartimento di Scienze Mediche, Università di Trieste, Trieste, Italy
3Istituto di Biochimica e Biologia Molecolare, Università di Amburgo, Amburgo, Germania
Contatto
Riccardo sola, email: riccardo.sola@phd.units.itRiferimento bibliografico
R. Sola et al.Characterization of Cetacean Proline-Rich Antimicrobial Peptides Displaying Activity against ESKAPE Pathogens
Int. J. Mol. Sci. 21, 7367 (2020)
DOI: 1422-0067/21/19/7367
Informazioni aggiornate al: 15.4.2021 alle ore 07:00
Contact: Webmaster - Dottorati Home pagina curata da: la Segreteria Dottorati di ricerca

Piazzale Europa, 1 - 34127 - Trieste, Italia -
Tel. +39 040 558 7111 - P.IVA 00211830328
C.F. 80013890324 - P.E.C. ateneo@pec.units.it



